¿Quieres estar al día? Suscríbete a nuestra newsletter. ¡1 E-mail cada 2 Meses! |
 |
1 de abril de 2022. La resistencia a los antibióticos provoca más de un millón de muertes al año en el mundo y se estima que esta cifra podría multiplicarse por diez en las próximas décadas. Alrededor de una décima parte de las muertes actuales se deben a infecciones por S. aureus resistente a meticilina. La proteína FtsZ de dicho patógeno, objeto del estudio, juega un papel central en la división celular, necesaria para la propagación de estas infecciones.
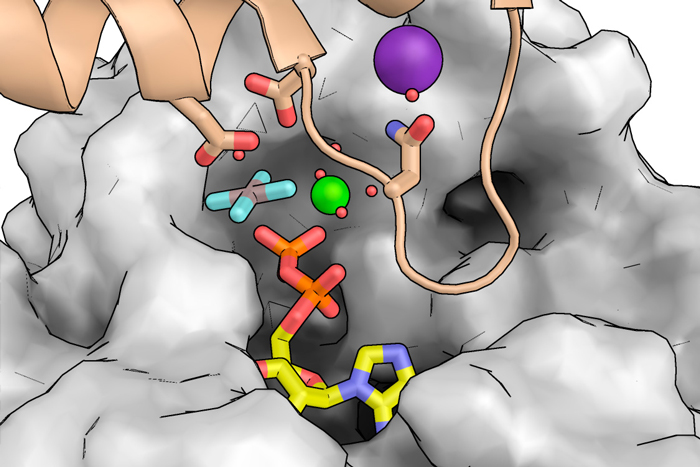
"La actividad de FtsZ en la división de la mayoría de las bacterias la convierte en blanco para descubrir nuevos antibióticos", señala Federico M. Ruiz, autor principal del trabajo. "FtsZ forma filamentos que, en presencia de cationes, hidrolizan el nucleótido GTP. Dichos filamentos crecen y decrecen de forma simultánea por extremos contrarios, lo que provoca un movimiento semejante al de una cinta transportadora. De este modo, en asociación con otras proteínas, FtsZ forma un anillo que permite la remodelación de la pared bacteriana y conduce a la división celular".
Esta investigación, codirigida por Carlos Fernández-Tornero y José Manuel Andreu del Centro de Investigaciones Biológicas Margarita Salas (CSIC), ha sido recientemente publicada en la revista PLoS Biology.
Usando la técnica de cristalografía de rayos X en el Sincrotrón ALBA y en el ESRF (Francia), el equipo científico obtuvo en alta resolución las estructuras de filamentos de FtsZ de S. aureus en complejo con diferentes cationes y análogos del nucleótido, incluidos los que mimetizan los estados basal y de transición del proceso de catálisis. Las estructuras proporcionan detalles atómicos del proceso por el que FtsZ coordina la hidrólisis del nucleótido GTP con cambios conformacionales esenciales para la dinámica de los filamentos formados por esta enzima. En combinación con un análisis mutacional y bioquímico, las estructuras obtenidas en las líneas de luz XALOC e ID23-1 han permitido identificar los aminoácidos de FtsZ que intervienen en este proceso.
"En conjunto —concluye Sonia Huecas, integrante del equipo investigador— estos hallazgos profundizan en el mecanismo íntimo de la división celular bacteriana y abren una vía para aplicaciones biomédicas".




