Sincrotrón ALBA
La estructura tridimensional de esta enzima ha estado obtenida gracias a la luz del Sincrotrón ALBA y ha permitido al equipo de la Universidad Autónoma de Barcelona identificar un nuevo mecanismo de regulación de una enzima que controla la vida media de las proteínas celulares. El estudio ha sido publicado en la revista Nature Communications.
Cerdanyola del Vallès, 31 de enero de 2019.
El control del tiempo de vida de las proteínas celulares es un mecanismo esencial para regular su actividad y para que funcione correctamente el ciclo celular.Este ciclo permite que las células puedan dividirse y así hacer que los tejidos crezcan y se regeneren.
El control de las diferentes etapas del ciclo celular está ejercido por diferentes tipos de proteínas que se van sustituyendo a medida que avanza el ciclo.
Las células han diseñado un mecanismo para marcar las proteínas que deben sustituirse para ser destruidas. Este proceso se denomina
ubiquitinación
, y la marca consiste en la formación de una cadena con la proteína
ubiquitina
.
Las proteínas marcadas (con
ubiquitina
) son eliminadas por una especie de gran recogida celular llamada
proteosoma
que se encarga de triturarlas.
La regulación de este proceso es muy importante para que el ciclo celular funcione correctamente, de lo contrario puede conllevar alteraciones en la proliferación celular que podrían conducir a procesos cancerígenos, donde los tejidos crecen sin control.
En el trabajo que el equipo del IBB (Instituto de Biotecnología y Biomedicina) de la UAB acaba de publicar en la revista
Nature Communications se
presenta la caracterización estructural y el descubrimiento de un nuevo mecanismo de regulación de una enzima que corta las cadenas de
ubiquitina
, que se llama USP25.
Esta enzima elimina las cadenas de
ubiquitina
de las proteínas marcadas y tiene un impacto importante en su función celular en evitar que sean degradadas por el
proteosoma
.
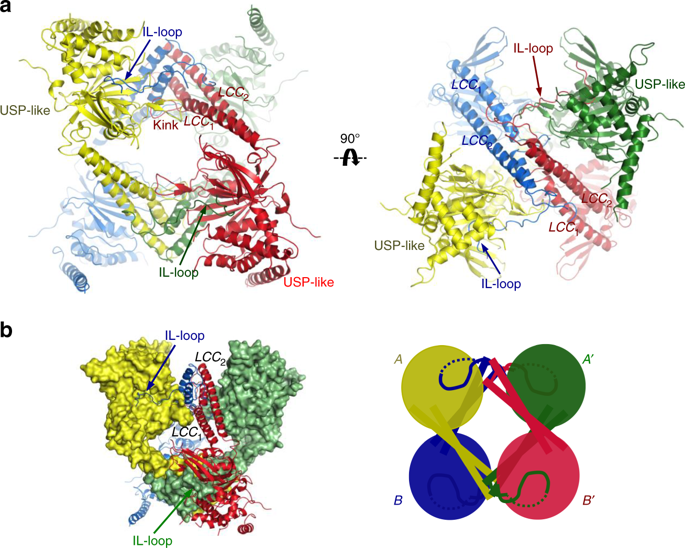
La estructura tridimensional de esta enzima, obtenida gracias a la cristalografía de proteínas y a la luz del Sincrotrón ALBA en la línea XALOC, ha permitido observar la presencia de un ensamblaje proteico con dos tipos diferentes de estructura cuaternaria: tetrámero y dímero.
Y lo que es más importante: el tetrámero es inactivo y el dímero es activo.
Por lo tanto, la alternancia entre estos dos ensamblajes regula la actividad de esta enzima y, de rebote, el tiempo de vida de importantes proteínas celulares.

USP25 es un homotetrámero formado por el ensamblaje de dos dímeros. a Representación del tetrámero de USP25. Cada subunidad está representada por un color diferente. Los dominios LLC y IL-loop están marcados. Diferentes orientaciones del tetrámero de USP25. b Representación mixta de la superficie y la cinta de la estructura del homotetrámero de USP25. Dibujo del conjunto del tetrámero formado por los dos homodímeros (A, A′ y B, B′). Imagen de Nature Communications.
Liu, B., Surena-Gómez, M., Zhen, Y., Amador, V., Reverter, D. A quaternary tetramer assembly inhibits the deubiquitinating activity of USP25. Nature Communications (2018 ). DOI: 10.1038 / s41467-018-07510-5.